DNA'nın yapısı
DNA yapısı, hem tek iplikli hem çift iplikli DNA'da çeşitli biçimler gösterir. Hücreler için DNA'nın yapısıyla ilişkili olan DNA'nın mekanik yapısı hücreler için önemli bir sorun yaratır. DNA'nın okunması veya ona bağlanmasıyla ilgili her hücresel süreç, onun tanınması, paketlenmesi veya değişime uğratılmasına etki edecek şekilde onun mekanik yapılarını da kullanır ya da değiştirir. DNA 'nın aşırı uzunluğunun (bir kromozomdaki DNA'nın uzunluğu 10 cm'yi bulabilir), onun sertliğinin ve sarmal yapısının bir sonucu olarak, hücre DNA'sının düzenlenebilmesi için histon gibi yapısal proteinler ve topoizomeraz ve helikaz gibi enzimler evrimleşmiştir. DNA'nın özellikleri onun moleküler yapısı ve dizisi ile yakından ilişkilidir. Özellikle DNA ipliklerini birbirine bağlayan hidrojen bağları ve elektronik etkileşimlerin, her bir iplikteki bağların kuvvetine kıyasla olan zayıflığı, bu ilişkide önemli bir rol oynar.
DNA'nın mekanik yapısını doğrudan ölçebilen deneysel teknikler nispeten yenidir ve çözelti içinde yüksek çözünümlü görüntüleme genelde zordur. Buna rağmen, bilimciler bu polimerin mekanik özellikleri hakkında büyük miktarda veri üretmişlerdir ve DNA'nın mekanik özelliklerinin hücresel süreçlere olan etkileri halen aktif olarak araştırılmakta olan bir konudur.
Çoğu hücrede bulunan DNA'nın uzunluk bakımından mikroskobik olduğunu belirtmek önemlidir—her bir insan kromozomundaki DNA birkaç santimetre uzunluğundadır. Dolayısıyla, hücreler DNA'yı içlerinde taşıyabilmek için onu sıkıştırmak veya "paketlemek" zorundadırlar. Ökaryotlarda, histon olarak adlandırılan makara gibi proteinler etrafından DNA'nın sarılması ile bu gerçekleşir. Bu DNA-protein kompleksinin daha da çok sıkıştırılması sonucu mitoz bölünme sırasında görülen kromozom yapıları meydana gelir.
Yapı belirlemesi
DNA yapılarının belirlenmesi nükleer manyetik rezonans veya X-ışını kristalogafisi teknikleri ile yapılır. A-DNA ve B-DNA'nın X-işini kırınım örüntülerinin ilk yayımlanan raporları Patterson transformlarına dayanan analizler kullanmış, bunlar buzağı timus DNA'sının yönlendirilmiş lifleri için sınırlı miktarda yapısal bilgi sağlamışlardı.[1][2] 1953'te Wilkins ve çalışma arkadaşları, bakteri ve alabalık sperm DNA'sının sulandırılmış (hidrate) ve yönlendirilmiş liflerindeki B-DNA'nın X-ışını kırınım ve saçılım örüntüleri için alternatif bir analiz yöntemini önerdiler, Bessel foksiyonlarının kareler toplamını kullanmak yoluyla.[3] `B-DNA biçimi' hücre içindeki şartlarda en yaygın olmakla beraber,[4] aslında bu iyi tanımlanmış bir üç boyutlu yapı (konformasyon) değil, canlı hücrelerin çoğunda bulunan sulanma seviyelerinde görülen bir DNA konformasyonlar ailesi veya konformasyonlar bulanık kümesidir (fuzzy set).[5] Bunlara karşılık gelen X-ışını kırınım ve saçılım örüntüleri, önemli oranda (>%20) bir düzensizlik içeren moleküler parakristallere özgündür,[6][7] ve bu yüzden standart analiz yöntemleri kullanılarak bunların yapıları çözülemez.
Buna karşın, Bessel fonksiyonlarının Fourier transformu[8] ve DNA moleküler modelleri kullanılarak yapılan standart analizler, A-DNA ve Z-DNA'nın X-ışını kırınım örüntülerinin analizinde rutin olarak hâlâ kullanılmaktadır.[9]
Baz çifti geometrisi
Bir baz çiftinin geometrisi 6 koordinat ile tanımlanabilir: yükselti, burulma, kayma, ötelenme, yatıklık ve yalpa (İngilizce rise, twist, slide, shift, tilt, ve roll). Bu değerler DNA molekülündeki her bir baz çiftinin uzaydaki konum ve doğrultusunu, sarmalda kendisinden bir evvelkine göreli olarak tam olarak tanımlar. Bunlar topluca molekülün sarmal yapısını tanımlarlar. Bir DNA molekülünde normal yapının bozulmuş olduğu bölgelerde bu değerler bozulmayı betimlemek için kullanılır.
Her bir baz çifti için aşağıdaki parametreler de tanımlanmıştır:[10][11][12]:
- Pervane burulması(Propeller twist)
- Aynı baz çiftindeki bir bazın düzleminin diğerinin düzlemine göre açısı
- Öteleme (Shift)
- Baz çifti düzleminde bir bazın ötekine göre kayma oranı, küçük oyuktan büyük oyuk doğrultusunda.
- Eğim(Tilt)
- Bu eksen etrafındaki dönme.
- Kayma (Slide)
- Baz çifti düzleminde bir iplikten ötekine doğru kayma.
- Baz çift düzlemini uzun ekseni etrafında dönmesi (Roll)
- rotation around this axis.
- Yükselme (Rise)
- sarmal ekseni boyunca ötelenme.
- Burulma (Twist)
- sarmal ekseni etrafında dönme.
- Hatve (Pitch)
- Sarmalda bir tam dönüşteki baz çifti sayısı
Yükselme ve burulma, sarmalın elliliğini ve hatvesini belirler. Diğer parametreler sıfıra eşit olabilir. Kayma ve ötelenme, B-DNA'da tipik olarak küçük değerlerdir ama A- ve Z-DNA'da büyük değerlere sahip olabilir. Yatıklık ve yalpa, ardışık baz çiftlerinin daha az paralel olmasına neden olur ve bunlar tipik olarak küçük değerlerdir. Bu parametreler hakkında bir şekil, 3DNA Web sitesinde görülebilir.
Bilimsel literatürde yatıklık (İngilizce "tilt") başka bir anlamda da kullanılabilir; ilk baz çifti ekseninin, sarmal eksenine olan diklikten olan sapması için de bu terim kullanılabilir. Bu anlam, ardışık iki baz çifti arasındaki kaymaya karşılık gelir ve sarmal-tabanlı koordinatlarda daha uygun olarak "eğim" (İng. "inclination") olarak belirtilir.
DNA sarmal geometrileri
Doğada üç DNA üç boyutlu yapısı (konformasyonu) olduğu düşünülmektedir, bunlar A-DNA, B-DNA, ve Z-DNA olarak adlandırılırlar. James D. Watson ve Francis Crick tarafından betimlenmiş olan "B" biçiminin hücrelerde hakim biçim olduğu görüşü yaygındır.[13] 23,7 Å genişliğindedir ve 10 baz çifti için 34 Å uzanır. Çifte sarmal her 10,4-10,5 baz çifti için bir tam dönüş yapar. Bu burulma sıklığı (sarmal hatvesi), her bir bazın komşularına yaptığı istifleme (İng. stacking) güçlerine bağlıdır.
Başka konformasyonlar da olasıldır; A-DNA, B-DNA, C-DNA, D-DNA[14], E-DNA[15], L-DNA(D-DNA'nın enatiomerik yapısı)[14], P-DNA[16], S-DNA, Z-DNA, v.s. tanımlanmıştır.[17] Gelecekte keşfedilebilecek DNA konformasyonları için F, Q, U, V, ve Y harfleri kalmıştır. Ancak bu biçimlerin çoğu suni olarak yaratılmış ve doğal olarak, biyoloji sistemlerde gözlemlenmemiştir.[18][19]
DNA'nın bir diğer yapı tipi, üç sarmallı DNA'dır.
A- ve Z-DNA
A ve Z-DNA, geometrileri bakımından birbirlerinden önemli derecede farklılık gösterirler ama ikisi de sarmal yapılıdırlar. A yapısı, sadece su kaybetmiş (dehidrate) DNA örneklerinde (kristalografik deneylerde olduğu gibi) ve belki DNA-RNA ipliklerinin hibrit eşleşmelerinde görülebildiği muhtemel sayılmaktadır. Hücrelerde DNA'nın metilasyona uğramış kısımları Z-DNA geometrisini sahip olabilir. Ayrıca bazı protein-DNA komplekslerinin Z-DNA yapıları oluşturduğuna dair deliller vardır.

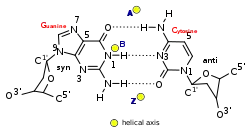
| Geometrik özellikleri | A-DNA | B-DNA | Z-DNA |
|---|---|---|---|
| Sarmal yön | sağ elli | sağ elli | sol elli |
| Tekrarlayan birim | 1 bp | 1 bp | 2 bp |
| Dönme/bç | 33.6° | 35.9° | 60°/2bp |
| Ortalama bç/dönme | 10.7 | 10.0 | 12 |
| Bç'nin eksene eğimi | +19° | -1.2° | -9° |
| Eksen boyunca yükselme/bç | 2.3 Å | 3.32 Å | 3.8 Å |
| Hatve/sarmal dönmesi | 24.6 Å | 33.2 Å | 45.6 Å |
| Ortalama pervane burulması | +18° | +16° | 0° |
| Glikosil açı | anti | anti | C: anti, G: syn |
| Şeker büzülmesi (İng. pucker) | C3'-endo | C2'-endo | C: C2'-endo, G: C2'-exo |
| Çap | 25.5 Å | 23.7 Å | 18.4 Å |
Süpersarımlı DNA
DNA'nın B biçimi her 10,4-10,5 bç bir tam dönüş yapar, torsiyon gerilimi olmayınca. Ancak, çeşitli biyolojik süreçler torsiyon gerilimi yaratır. Aşırı veya eksik sarmal gerilimli bir DNA parçasına pozitif veya negatif "süpersarımlı" olarak değinilir. Hücrelerdeki DNA tipik olarak negatif süpersarımlıdır, onun böyle olması ikili sarmalın çözülüp (eriyip) transkripsiyonun olmasını sağlar.
Sarmal olmayan biçimler
DNA'nın sarmal olmayan biçimleri de betimlenmiştir, örneğin yan yana ve üçlü sarmal biçimleri. Tek iplikli DNA, DNA ikilenmesi sırasında veya ısı ile DNA ipliklerinin ayrışması sonucu meydana gelir.
DNA bükülmesi
DNA göreceli olarak rijit bir polimer sayılır, Solucanvari zincir olarak modellenir. Üç önemli serbestlik derecesi vardır: bükülme, burulma ve sıkışma. Bunların her biri DNA'nın hücre içindeki yeteneklerine belli sınırlamalar getirir. Burulma/torsiyonal tutukluğu, DNA'nın halkasallaşmasına ve DNA'ya bağlanan proteinlerin birbirlerine göreceli doğrultusuna etki eder. Bükülme/eksensel tutukluğu DNA'nın sarılmasına, onun halkasallaşmasına ve proteinlerle etkileşimine etki eder. Sıkışma (kompresyon)/uzama ise yüksek gerilme hâli dışında nispeten önemsizdir.
Süreğenlik uzunluğu/Eksensel katılık
| Dizi | Süreğenlik uzunluğu /baz çifti |
|---|---|
| Rastegele | 154±10 |
| (CA)tekrarı | 133±10 |
| (CAG)tekrarı | 124±10 |
| (TATA)tekrarı | 137±10 |
Çözeltideki DNA'nın rijit bir yapısı yoktur, termal titreşimler ve su molekülleri ile çarpışmalar nedeniyle sürekli değişen bir konformasyona sahiptir ve bu yüzden rijitliğin klasik ölçümleri mümkün değildir. Dolayısıyla DNA'nın bükülmezliği onun süreğenlik uzunluğu ile ölçülür, bunun tanımı şöyledir:
- "polimerin zamana-bağllı ortalama doğrultusunun e faktörü ile bağıntısız (ilintisiz) olduğu DNA uzunluğu"
Bu değer atomik kuvvet mikroskobu ile farklı uzunlukta DNA moleküllerini görüntüleyerek doğrudan ölçülebilir. Sulu çözeltilerde ortalama süreğenlik uzunluğu 46-50 nm veya 140-150 baz çiftidir (DNA'nın çapı 2 nm'dir), ama bu değer büyük çeşitlilik gösterebilir. Bu tanıma göre DNA orta derecede rijit bir molekül sayılır.
DNA'nın belli bir kısmının süreğenlik uzunluğu kısmen onun dizisine bağlı olduğu için büyük bir varyasyon gösterebilir. Bu varyasyon büyük ölçüde baz istiflenme enerjisinde ve küçük ve büyük oluklara uzanan bazlardan kaynaklanır.
DNA bükülmesinin modelleri
| Basamak | İstiflenme ΔG /kcal mol−1 |
|---|---|
| T A | -0.19 |
| T G or C A | -0.55 |
| C G | -0.91 |
| A G or C T | -1.06 |
| A A or T T | -1.11 |
| A T | -1.34 |
| G A or T C | -1.43 |
| C C or G G | -1.44 |
| A C or G T | -1.81 |
| G C | -2.17 |
DNA'nın entropik esnekliği standart polimer fiziği modelleri (Kratky-Porod solucan benzeri zincir modeli gibi) ile dikkate değer derecede uyumludur. Solucan benzeri zincir modelinin öngördüğü gibi, küçük (picoNewton-altı) kuvvetlerde Hooke kanunu tarafından betimlenir. Ancak, süreğenlik uzunluğunundan kısa uzunlukta DNA parçalarında bükülme kuvveti yaklaşık sabittir ve davranışı, solucan benzeri zincir öngörülerinden sapma gösterir. Bunun sonucu olarak küçük DNA molekülleri kolay halkalaşır ve DNA'da çok bükük kısımların bulunma olasılığı daha yüksektir.
Bükülme tercihleri
DNA moleküllerinin bükülmesine tercihli yönler vardır, yani DNA anizotropik bükülme gösterir. Bunun nedeni DNA dizisini oluşturan bazların özellikleridir; rastgele bir dizinin tercihli bir bükülme yönü yoktur.
Tercihli DNA bükülmesi, her bazın komşusu üzerinde istiflenmesinin stabilitesi tarafından belirlenir. Eğer kararsız baz istiflenmesi DNA sarmalının hep aynı tarafında yer alırsa DNA o yöne doğru bükülür. Bükülme açısı artınca sterik engeller ve bazların birbirine göre yuvarlanması da bükülmede rol oynar, özellikle küçük olukta. A ve T bazları bükülmelerin iç tarafında küçük olukta tercihen bulunurlar. Bu etki özellikle DNA-protein bağlanması sonucu sıkı bükülmenin oluştuğu yerlerde görülür, nükleozom taneciklerinde olduğu gibi. Yukarıdaki tabloda baz çarpıtmalarına (distorsiyonlarına) bakınız.
İstisnai bükülme tercihi olan DNA molekülleri içsel olarak büküktürler. Bu olgu ilk defa tripanozomlardaki kinetoplast DNA'sında gözlemlenmiştir. Buna neden olan tipik DNA dizileri 4-6 T ve A bazından oluşan bölümler ve bu bölümleri DNA'nın hep aynı tarafındaki küçük oluğa rastlatacak şekilde aralarda G ve C-zengini bölümlerdir. Örneğin:
| | | | | | G A T T C C C A A A A A T G T C A A A A A A T A G G C A A A A A A T G C C A A A A A A T C C C A A A C
Bu içsel olarak bükük yapıda baz çiftlerindeki 'pervane burulması' meydan gelir, yani baz basamakları arasında anormal çatallaşmış Hidrojen bağları oluşabilir. Yüksek sıcaklıklarda bu yapı ve onun neden olduğu içsel büküklük kaybolur.
Anizotropik olarak bükülen her DNA'nın süreğenlik uzunluğu ortalamadan daha fazladır ve eksensel bükülmezliği daha çoktur, yani rastgele bükülme olasılığı daha düşüktür.
DNA halkalaşması
DNA halkalaşması molekülün hem eksensel (bükülme) sertliği hem de torsiyonal (dönel) sertliği ile ilişkilidir. Bir DNA molekülünün başarılı bir şekilde halkalaşabilmesi için tam halka olabilecek kadar uzun olması gerekir; buna ilaveten, kovalent bağların oluşabilmesi için uçtaki bazların doğru açıya sahip olması gerekir, bunun için de molekülde doğru sayıda baz bulunması gerekir. DNA halkalaşması için optimal uzunluk 400 baz çiftidir (136 nm uzunluk), DNA sarmalındaki dönmelerin sayısı tam sayı olmak zorundadır. Dönme sayısı tam sayı değilse halkalaşmak için hatırı sayılır bir enerji bariyeri yaratır; örneğin 10,4 x 30 = 312 baz çiftli bir molekül 10,4 x 30,5 ≈ 317 baz çiftli bir molekülde yüzlerce kere daha hızlı halkalaşır.
DNA uzaması
Uzun DNA parçaları gerilim altında entropik olarak elastiklik gösterirler. DNA çözeltideyken, solventte bulunan enerjiyle ilişkili olarak sürekli yapısal varyasyonlar geçirir. Bunun nedeni, moleküldeki ısıl (termal) titreşimler ve, buna ek olarak, su molekülleri ile olan sürekli çarpışmalardır. Entropik nedenlerden dolayı, sıkışık ama gevşek olan yapılar ısının bu etkilerine daha duyarlıdır, uzamış yapılara kıyasla, ve bu nedenle DNA molekülleri evrensel olarak karışık ve gevşek yapılara sahiptir. Bu nedenle tek bir DNA molekülü kuvvet etkisiyle uzayıp düzleşir. Optik cımbız kullanarak DNA'nın entropik uzama davranışı incelenmiş ve fizyolojik sıcaklıklarda Kratky-Porod solucan benzeri zincir modelindeki gibi davrandığı bulunmuştur.
Yeterli gerilim ve pozitif buru (tork) etkisi altında DNA bir faz değişmesi (evre geçişi) gösterir, bazlar dışarı, fosfatlar içeri döndüğü öne sürülmüştür. Bu aşırı gerilmiş DNA için önerilen yapı "P-biçimli DNA" olarak adlandırılmıştır, DNA'nın yapısı daha bilinmezken bu yapıyı önermiş olan Linus Pauling'e atfen.[16]
DNA'nın sıkıştırılıncaki mekanik yapısı betimlenmemiştir, polimerin sıkıştırıcı kuvvet altın bükülmesini engellemenin teknik zorluklarından dolayı.
DNA ergimesi
| Basamak | Ergime ΔG /Kcal mol−1 |
|---|---|
| T A | -0.12 |
| T G or C A | -0.78 |
| C G | -1.44 |
| A G or C T | -1.29 |
| A A or T T | -1.04 |
| A T | -1.27 |
| G A or T C | -1.66 |
| C C or G G | -1.97 |
| A C or G T | -2.04 |
| G C | -2.70 |
DNA ergimesi, ikili sarmalın iplikleri arasındaki etkileşimlerin bozulup iki ipliğin ayrışması sürecidir. Bu bağlar zayıftır, hafif ısıtma, enzimler veya fiziksel kuvvet ile kolayca kırılırlar. DNA erigimesi tercihli olarak DNA üzerinde belli noktalarda meydana gelir.[20] T ve A zengini diziler, C ve G zengini dizilere kıyasla daha kolay ergir. Belli baz basamakları da DNA ergimesine daha müsaittir, özellikle TA basmaklar ve TG baz basamakları.[21] Bu mekanik özellikler, çoğu genin baş tarafında bulunan TATAA dizisinin varlığını açıklar; bu dizinin kolay ergiyebilir olması, RNA polimerazın transkripsiyona başlamak için DNA ipliklerini ayırmasını sağlar.
Hafif ısıtma ile ipliklerin ayrılması, polimeraz zincir tepkimesinde (PCR) olduğu gibi, eğer molekül 10.000 baz çiftinden (10 kilobaz çifti veya 10 kbç) küçük ise basittir. DNA ipliklerinin birbirine sarılmış olması uzun DNA ipliklerinin birbirnden ayrılmasını güç kılar. Hücre bu sorunun üstesinden gelmek için topoizomerazlarla beraber çalışan DNA ergitme enzimleri (helikazlar) kullanır. Topoizomerazlar iki iplikten birinin şeker-fosfat omurgasını kimyasal olarak keserek DNA'nın öbür ipliği etrafında dönmesini sağlar. Helikazlar ipliklere çözerek DNA polimeraz gibi dizi okuyucu enzimlerin ilerlemesini sağlar.
DNA topolojisi
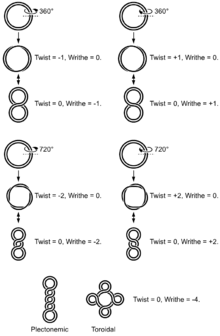
Hcredeki çoğu DNA topolojik olarak kısıtlanmıştır. DNA ya kapalı halka şeklindedir (prokaryotlardaki plazmitler gibi) ya da çok uzun moleküllerdir ki, bunlar, düşük difüzyon katsayısı yüzünden bunlar fiilen topolojik olarak kapalı bölgeler meydana getirir. DNA'nın lineer kısımları da çoğu zaman membranlara bağlı proteinler tarafından bağlıdır ve bunun sonucu topolojik anlamda kapalı halkalar oluşur.
Francis Crick DNA süpersarımlarında bağlantı sayısının önemini ilk öneren kişilerden olmuştur. 1976'da yayımlanan bir makalede, Crick problemi dile getirmiştir[22]
DNA topolijisinin analizinde üç değer kullanılır:
- L = Bağlantı sayısı (İng. linking number) Bir DNA ipliğinin öbürü etrafında kaç kere döndüğünün sayıs. Kapalı bir halka için bu bir tam sayıdır, kapalı bir topolojik bölge için de bu sabittır.
- T = burulma (İng. twist) İki iplikli DNA sarmalındaki toplam dönüş sayısı. Normalde bu sayı DNA çözeltide serbest bulunduğu zamanki dönüş sayısına eşittir, yani, baz sayısı/10,4
- W= burkulma
- L = T + W ve ΔL = ΔT + ΔW
Kapalı bir topolojik bir bölgede T'deki bir değişme, W'deki bir değişme ile dengelenmek zorundadır ve bu ilişkinin tersi de doğrudur. Bunun sonucu DNA'nın üst düzey yapısı oluşur. Burkulma sayısı 0 olan halkasal bir DNA molekülü halkasaldır. Eğer molekülün burulması süpersarım yapılarak azaltılır veya artırılırsa, burkulma da uygun şekilde değişir, öyle ki molekülde simitsi veya çubuksu süpersarmal bir sarım meydana gelir.
Eğer çift iplikli sarmal DNA'nın uçları birleştirilip bir halka oluşursa iplikler topolojik anlamda düğümlenmiş olurlar. bu demektir ki, iplikler kesilmeden birbirlerinden ayrılamazlar; ısıtmak DNA'yı ergitse dahi, iplikler gene de birbirleri etrafında sarılı durumda kalırlar. Topolojik olarak bağlı ipliklerin düğümünün çözülmesi için topoziomeraz denen enzimler gereklidir. Bu enzimler halkasal DNA'nın düğümlü halini çözmek için ipliklerin biri veya ikisini birden keseler, öyle ki başka bir tek veya iki iplikli DNA parçası onun içinden geçebilsin. Bu düğüm çözümü halkasal DNA'nın (veya topolojik olarak benzer şekilde kısıtlanmış doğrusal DNA'nın) ikilenmesi ve çeşitli rekombinasyon tiplerinde gereklidir.
Bağlantı sayısı paradoksu
Yıllar boyunca ökaryotik genomlardaki süpersarılımın kaynağı gizemli kalmıştır. Bu topolojik bilmece bazılarınca "bağlantı sayısı paradoksu" olarak adlandırılmıştı.[23] Ancak, nükleozomun yapısı çözülünce ve onun etrafında sol-elli aşırı burulmuş bir DNA olduğu görülünce bu "paradoks" çözülmüştür.[24][25]
Ayrıca bakınız
Kaynakça
- Franklin, R.E. and Gosling, R.G. received 6 March 1953. Acta Cryst. (1953). 6, 673: The Structure of Sodium Thymonucleate Fibres I. The Influence of Water Content.; also Acta Cryst. 6, 678: The Structure of Sodium Thymonucleate Fibres II. The Cylindrically Symmetrical Patterson Function.
- Franklin, Rosalind (1953). "Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G" (PDF). Nature. Cilt 171. ss. 740-741. doi:10.1038/171740a0. PMID 13054694. 3 Ocak 2011 tarihinde kaynağından (PDF) arşivlendi. Erişim tarihi: 3 Ağustos 2009.
- Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. (1953). "Molecular Structure of Deoxypentose Nucleic Acids" (PDF). Nature. Cilt 171. ss. 738-740. doi:10.1038/171738a0. PMID 13054693. 13 Mayıs 2011 tarihinde kaynağından (PDF) arşivlendi. Erişim tarihi: 3 Ağustos 2009.
- Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). "Polymorphism of DNA double helices". J. Mol. Biol. 143 (1). ss. 49-72. doi:10.1016/0022-2836(80)90124-2. PMID 7441761.
- Baianu, I.C. (1980). "Structural Order and Partial Disorder in Biological systems". Bull. Math. Biol. 42 (4). ss. 464-468. doi:10.1016/0022-2836(80)90124-2.
- Hosemann R., Bagchi R.N., Direct analysis of diffraction by matter, North-Holland Publs., Amsterdam – New York, 1962
- Baianu I.C., X-ray scattering by partially disordered membrane systems, Acta Cryst. A, 34 (1978), 751–753.
- Bessel functions and diffraction by helical structures
- "X-Ray Diffraction Patterns of Double-Helical Deoxyribonucleic Acid (DNA) Crystals". 24 Temmuz 2009 tarihinde kaynağından arşivlendi. Erişim tarihi: 3 Ağustos 2009.
- Dickerson RE (1989). "Definitions and nomenclature of nucleic acid structure components". Nucleic Acids Res. 17 (5). ss. 1797-1803. doi:10.1093/nar/17.5.1797. PMID 2928107.
- Lu XJ, Olson WK (1999). "Resolving the discrepancies among nucleic acid conformational analyses". J Mol Biol. 285 (4). ss. 1563-1575. doi:10.1006/jmbi.1998.2390. PMID 9917397.
- Olson WK, Bansal M, Burley SK, Dickerson RE, Gerstein M, Harvey SC, Heinemann U, Lu XJ, Neidle S, Shakked Z, Sklenar H, Suzuki M, Tung CS, Westhof E, Wolberger C, Berman HM (2001). "A standard reference frame for the description of nucleic acid base-pair geometry". J Mol Biol. 313 (1). ss. 229-237. doi:10.1006/jmbi.2001.4987. PMID 11601858.
- Richmond; ve diğerleri. (2003). "The structure of DNA in the nucleosome core". Nature. Cilt 423. ss. 145-150. doi:10.1038/nature01595. PMID 12736678.
- Hayashi G, Hagihara M, Nakatani K (2005). "Application of L-DNA as a molecular tag". Nucleic Acids Symp Ser (Oxf). Cilt 49. ss. 261-262. PMID 17150733.
- Vargason JM, Eichman BF, Ho PS (2000). "The extended and eccentric E-DNA structure induced by cytosine methylation or bromination". Nature Structural Biology. Cilt 7. ss. 758-761. doi:10.1038/78985.
- Allemand; ve diğerleri. (1998). "Stretched and overwound DNA forms a Pauling-like structure with exposed bases". PNAS. Cilt 24. ss. 14152-14157. doi:10.1073/pnas.95.24.14152. PMID 9826669.
- "List of 55 fiber structures". 26 Mayıs 2007 tarihinde kaynağından arşivlendi. Erişim tarihi: 3 Ağustos 2009.
- Bansal M (2003). "DNA structure: Revisiting the Watson-Crick double helix". Current Science. 85 (11). ss. 1556-1563.
- Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z". Acta Cryst. Cilt D59. ss. 620-626. doi:10.1107/S0907444903003251.
- Breslauer KJ, Frank R, Blöcker H, Marky LA (1986). "Predicting DNA duplex stability from the base sequence". PNAS. 83 (11). ss. 3746-3750. PMID 3459152.
- Richard Owczarzy (28 Ağustos 2008). "DNA melting temperature - How to calculate it?". High-throughput DNA biophysics. owczarzy.net. 30 Nisan 2015 tarihinde kaynağından arşivlendi. Erişim tarihi: 2 Ekim 2008.
- Crick FH (1976). "Linking numbers and nucleosomes". Proc Natl Acad Sci USA. 73 (8). ss. 2639-43. doi:10.1073/pnas.73.8.2639. PMID 1066673.
- Prunell A (1998). "A topological approach to nucleosome structure and dynamics: the linking number paradox and other issues". Biophys J. 74 (5). ss. 2531-2544. PMID 9591679.
- Luger K, Mader AW, Richmond RK, Sargent DF, Richmond TJ (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648). ss. 251-260. doi:10.1038/38444. PMID 9305837.
- Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ (2002). "Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution". Journal of Molecular Biology. 319 (5). ss. 1097-1113. doi:10.1016/S0022-2836(02)00386-8. PMID 12079350.
Dış bağlantılar
- MDDNA: Structural Bioinformatics of DNA (DNA'nın yapısal biyoenfromatiği)
- DNAlive: DNA'nın fiziksel özelliklerinin hesaplanması için bir Web arayözü. Sonuçların UCSC Genom tarayıcısı ile ilişkilendirilmesine olanak sağlamaktadır.
- DiProDB: Dinükleotit özellikleri veritabanı18 Temmuz 2019 tarihinde Wayback Machine sitesinde arşivlendi.. Bu veritabanı dinükleotitlerin termodinamik, yapısal ve diğer özelliklerinin toplanması ve analiz edilmesi amacıyla yaratılmıştır.
