Dış grup (kladistik)
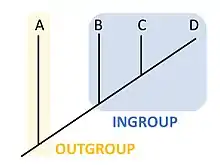
Kladistik ya da filogenetikte, bir dış grup[1] incelenen organizma grubu içindeki evrimsel ilişkileri belirlerken daha uzaktan ilişkili olan ve referans grubu olarak kullanılan organizmalar grubudur. Dış grup, iç grup için bir karşılaştırma noktası olarak kullanılır ve özel olarak da filogeninin kökünün olmasını sağlar. Özellik değişikliğinin yönü yalnızca kökü olan bir filogeni sayesinde belirlenebildiği için dış grubun seçimi bir filogeni içinde özelliklerin evrimini anlayabilmek için gereklidir.[2]
Tarihçe
Her ne kadar dış grup kavramı kladistiğin ilk günlerinden beri kullanılmış olsa da "dış grup" terimi 1970'lerin başlarında Amerikan Doğa Tarihi Müzesi'nde kullanılmaya başlandığı düşünülmektedir.[3]
Dış grup seçimi
Seçilen dış grubun iç grup ile, iç grubun kendi içinde olduğundan daha az ilişkili olduğu varsayılır. Bu ilişkilerden çıkarılan evrimsel sonuç dış grup türlerinin iç grup ile olan ortak atalarının iç grubun ortak atasından daha eski olduğudur. Dış grup seçimi bir filogeninin topolojisini değiştirebilir.[4] Dolayısıyla filogenetikçiler kladistik analizde birden fazla dış grup kullanırlar. Çoklu dış grup kullanımının tercih edilmesinin nedeni zayıf dış grup adaylarının seçilmesine karşın iç grubun monofiletikliği varsayımını sınayabilmek için daha sağlam bir filogeni oluşturulmasına olanak vermesidir.[3][5][6]
Bir dış grup olarak değerlendirilebilmesi için bir taksonun şu özellikleri sağlaması gerekir:
- İç grubun bir üyesi olmamalı,
- İç grupla anlamlı bir karşılaştırma yapılabilmesi için iç grup ile ilişkili olmalı.
Dolayısıyla uygun bir dış grup filogenetik araştırmaya konu olan kladın açık bir şekilde dışında yer almalıdır. İç grubun içine yerleştirilebilecek bir dış grup filogeninin kökü için kullanıldığında filogenetik ilişkilerin ve özelliklerin evrimi konusunda doğru olmayan sonuçlara yol açacaktır.[7] Ancak dış grubun iç gruba olan ilişkisinin optimal düzeyi filogenetik analizin derinliğine bağlıdır. Çok belirgin olmayan nüansların incelenmesinde iç gruba çok yakın akraba bir dış grubun seçilmesi daha uygunken gereksiz yere çok uzak bir dış grubun seçilmesi en son ortak ata nedeniyle olan doğrudan evrimsel ilişkinin yakınsak evrim ile karıştırılmasına neden olabilir.[8][9] Daha basit filogenetik için örneğin bir cins içindeki kladın evrimsel ilişkilerini çözümlemede dış grup kardeş kladın bir üyesi olabilir.[10] Ancak daha derin filogenetik analiz için daha az ilişkili taksonlar kullanılabilir. Örneğin, Jarvis vd. (2014) kuş filogenisinin erken dalları için dış grup olarak insanları ve timsahları kullanmışlardır.[11] Moleküler filogenetikte ikinci gerekliliği karşılamak genel olarak dış grubun DNA ya da protein dizilerinin iç grubun dizileri ile başarılı bir şekilde hizalanmasıdır. Her ne kadar maksimum global sadelik ile dış grupları belirlemek için algoritmik yaklaşımlar bulunsa da genellikle bazı özellik durumlarının sürekli ve nicel doğasını yansıtamama nedeniyle sınırlara sahiptirler.[12] Özellik durumları atadan gelen ya da gelişmiş olan ve filogenetik ağacın dallanma örüntüsünün kurulumunu etkileyen niteliklerdir.[13]
Örnekler
Ayrıca bakınız
- Apomorfi, bir organizmanın gelişmiş özelliği
- Kardeş grup, aiç gruba yakından ilişkili olabilen grup
- Plesiomorfi, bir organizmanın atadan kalma özelliği
- İlkel (filogenetik), atadan kalma özellikler için kullanılan terim
Kaynakça
- Grimaldi, David; Engel, Michael S.; Engel, Michael S. (16 Mayıs 2005). Evolution of the Insects. ISBN 9780521821490.
- Farris, J. S. (1982). "Outgroups and Parsimony". Systematic Biology. 31 (3). ss. 328-334. doi:10.1093/sysbio/31.3.328. ISSN 1063-5157.
- Nixon, Kevin; Carpenter, James (Aralık 1993). "On Outgroups". Cladistics. 9 (4). ss. 413-426. doi:10.1111/j.1096-0031.1993.tb00234.x.
- Giribet, G.; Ribera, C. (Haziran 1998). "The position of arthropods in the animal kingdom: a search for a reliable outgroup for internal arthropod phylogeny". Molecular Phylogenetics and Evolution. 9 (3). ss. 481-488. doi:10.1006/mpev.1998.0494. PMID 9667996.
- Barriel, V.; Tassy, P. (Haziran 1998). "Rooting with Multiple Outgroups: Consensus Versus Parsimony". Cladistics. 14 (2). ss. 193-200. doi:10.1111/j.1096-0031.1998.tb00332.x.
- de la Torre-Barcena, Jose Eduardo; Kolokotronis, S.O.; Lee, Ernest; Stevenson, Dennis; Brenner, Eric; Katari, Manpreet; Coruzzi, Gloria; DeSalle, Rob (2009). "The Impact of Outgroup Choice and Missing Data on Major Seed Plant Phylogenetics Using Genome-Wide EST Data". PLOS ONE. 4 (6). ss. e5764. doi:10.1371/journal.pone.0005764. PMC 2685480 $2. PMID 19503618.
- Maddison, Wayne (1984). "Outgroup Analysis and Parsimony" (PDF). Systematic Zoology. 33 (1). ss. 83-103. doi:10.2307/2413134. JSTOR 2413134.
- Wilberg, Eric W. (1 Temmuz 2015). "What's in an Outgroup? The Impact of Outgroup Choice on the Phylogenetic Position of Thalattosuchia (Crocodylomorpha) and the Origin of Crocodyliformes". Systematic Biology. 64 (4). ss. 621-637. doi:10.1093/sysbio/syv020. ISSN 1063-5157. PMID 25840332. Geçersiz
|doi-access=free(yardım) - O'Brien, Michael J.; Lyman, R.Lee; Saab, Youssef; Saab, Elias; Darwent, JOohn; Glover, Daniel S. (2002). "Two Issues in Archaeological Phylogenetics: Taxon Construction and Outgroup Selection". Journal of Theoretical Biology. 215 (2). ss. 133-150. doi:10.1006/jtbi.2002.2548. PMID 12051970.
- Baum, David A.; Smith, Stacey D. (2013). Tree Thinking: An Introduction to Phylogenetic Biology. Roberts. s. 175. ISBN 978-1-936221-16-5.
- Jarvis, E. (Aralık 2014). "Whole-genome analyses resolve early branches in the tree of life of modern birds". Science. 346 (6215). ss. 1320-1331. doi:10.1126/science.1253451. PMC 4405904 $2. PMID 25504713.
- Stevens, P. F. (1991). "Character States, Morphological Variation, and Phylogenetic Analysis: A Review". Systematic Botany. 16 (3). ss. 553-583. doi:10.2307/2419343. JSTOR 2419343.
- Rineau, Valentin; Grand, Anaïs; Zaragüeta, René; Laurin, Michel (1 Mayıs 2015). "Experimental systematics: sensitivity of cladistic methods to polarization and character ordering schemes". Contributions to Zoology. 84 (2). ss. 129-148. doi:10.1163/18759866-08402003. Geçersiz
|doi-access=free(yardım) - Prado-Martinez, Javier; Marques-Bonet, Tomas (2013). "Great ape genetic diversity and population history". Nature. 499 (7459). ss. 471-475. doi:10.1038/nature12228. PMC 3822165 $2. PMID 23823723.
- Murphy, William; Pringle, Thomas; Crider, Tess; Springer, Mark; Miller, Webb (2007). "Using genomic data to unravel the root of the placental mammal phylogeny". Genome Research. 17 (4). ss. 413-421. doi:10.1101/gr.5918807. PMC 1832088 $2. PMID 17322288.
- Cameron, Chris; Garey, James; Swalla, Billie (2000). "Evolution of the chordate body plan: New insights from phylogenetic analyses of deuterostome phyla". PNAS. 97 (9). ss. 4469-4474. doi:10.1073/pnas.97.9.4469. PMC 18258 $2. PMID 10781046.
- Matthews, Sarah; Donoghue, Michael (1999). "The Root of Angiosperm Phylogeny Inferred from Duplicate Phytochrome Genes". Science. 286 (5441). ss. 947-950. doi:10.1126/science.286.5441.947. PMID 10542147.
