Ökaryotik transkripsiyon
Ökaryotik transkripsiyon , ökaryotik hücrelerin DNA'da depolanan genetik bilgiyi RNA replika birimlerine kopyalamak için kullandıkları ayrıntılı bir işlemdir. Gen transkripsiyonu hem ökaryotik hem de prokaryotik hücrelerde görülür. Tüm farklı RNA tiplerinin transkripsiyonunu başlatan prokaryotik RNA polimerazının aksine, ökaryotlardaki (insanlar dahil) RNA polimerazlar, her biri farklı bir gen tipini kodlayan üç varyasyona sahiptir. Bir ökaryotik hücre, transkripsiyon ve translasyon işlemlerini ayıran bir çekirdeğe sahiptir. Ökaryotik transkripsiyon, DNA'nın nükleozomlara ve daha yüksek dereceli kromatin yapılarına paketlendiği çekirdeğin içinde meydana gelir. Ökaryotik genomun karmaşık oluşu, komplex ve çok çeşitli bir gen anlatım kontrol mekanizmasının varlığını gerektirir.
Özet
Transkripsiyon, bir DNA zincirinde depolanan genetik bilginin, taşınabilir bir tamamlayıcı RNA zincirine kopyalanması işlemidir. Ökaryotik transkripsiyon hücrenin çekirdeğinde gerçekleşir ve üç ardışık aşamada ilerler: başlama, uzama ve sonlandırma.[1] Bu karmaşık reaksiyonu katalize eden transkripsiyonel makine, temelinde üç çeşit , birden fazla alt birime sahip olan, RNA polimeraza sahiptir. RNA polimeraz I, ribozomun yapısal RNA' larının transkripsiyonundan sorumludur.
Protein kodlayan genler, bilgiyi DNA'dan protein sentezine taşıyan haberci RNA'lara (mRNA'lara) transkribe edilir. mRNA'lar büyük bir çeşitliliğe sahip olmasına rağmen, hücrede üretilen en bol RNA türü değildir. Kodlama yapmayan RNA'lar bir hücrenin transkripsiyonel çıktısının büyük çoğunluğunu oluşturur.[2] Bu kodlamayan RNA'lar, çeşitli önemli hücresel fonksiyonları yerine getirir.
RNA polimeraz
Ökaryotların her biri farklı rol ve özelliklere sahip üç nükleer RNA polimerazı vardır
| isim | yer | Ürün |
| RNA Polimeraz I (Pol I, Pol A) | çekirdekçik | daha büyük ribozomal RNA ( rRNA ) ( 28S , 18S , 5.8S ) |
| RNA Polimeraz II (Pol II, Pol B) | çekirdek | haberci RNA ( mRNA ), çoğu küçük nükleer RNA (small-nuclear RNA/ snRNA ), küçük müdehalaci RNA (small-interfering RNA siRNA'lar ) ve mikroRNA (Mikro RNA/ miRNA ). |
| RNA Polimeraz III (Pol III, Pol C) | çekirdek (ve muhtemelen nükleolus- nükleoplazm arayüzü) | transfer RNA ( tRNA ), diğer küçük RNA'lar (küçük 5S ribozomal RNA (5s rRNA dahil) , snRNA U6, sinyal tanıma partikül RNA (Signal recognition particle RNA/ SRP RNA) ve diğer kararlı kısa RNAlar dahil |
RNA polimeraz I (Pol I), 5S hariç tüm rRNA genlerinin transkripsiyonunu katalize eder. Bu rRNA genleri, tek bir transkripsiyonel birim halinde kontrol edilir. Bu öncül daha sonra üç rRNA'ya işlenir: 18S, 5.8S ve 28S. rRNA genlerinin transkripsiyonu çekirdekçikte olur.[3] Çekirdekçikte rRNA transkriptleri ribozom oluşturmak amaçlı proteinler ile birleşirler.[4]
RNA polimeraz II (Pol II), tüm mRNA'ların, bazı snRNA'ların, siRNA'ların ve tüm miRNA'ların transkripsiyonundan sorumludur. Birçok Pol II transkripti, geçici olarak, olgun RNA'lar üretmek için işlenen tek iplikli öncü RNA'lar (öncül RNA) olarak bulunur. Örneğin, öncül mRNA'lar, protein translasyonu için, nükleer por boyunca, sitoplazmaya çıkmadan önce kapsamlı bir şekilde işlenir.
RNA polimeraz III (Pol III), tRNA'lar, 5S rRNA, U6 snRNA, SRP RNA ve ribonükleaz P RNA gibi diğer kararlı kısa RNAlar dahil olmak üzere kodlamayan küçük RNA'ları transkribe eder.[5]
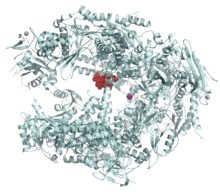
RNA Polimeraz I, II ve III, sırasıyla 14, 12 ve 17 alt birim içerir.[6] Üç ökaryotik polimerazın hepsinde, E. coli RNA polimerazının β, β ', α I , α II ve ω alt birimleriyle benzerlik gösteren beş temel alt birim mevcuttur. Aynı ω benzeri bir alt birim (RBP6), üç ökaryotik polimerazın tümü tarafından kullanılırken, aynı a-benzeri alt birimler Pol I ve III tarafından kullanılır. Üç ökaryotik polimeraz arasında dört ortak alt birim vardır. Kalan alt birimler, her RNA polimeraza özgündür. Pol II ve Pol III'te Pol II'ye göre fazladan bulunan ek alt birimler, Pol II transkripsiyon faktörlerine homologtur.
RNA polimeraz I [7] ve II [8] nin kristal yapıları alt birimler arasındaki etkileşimleri ve ökaryotik transkripsiyonun moleküler mekanizmasını atomik detaylarla anlama fırsatı sunmuştur.
RNA polimeraz II'nin en büyük alt birimi olan RPB1'in karboksil terminal domaini (carboxyl terminal domain/ CTD), Pol II transkriptlerinin sentezi ve işlenmesi için gerekli mkanizmanın bir araya getirilmesinde önemli bir rol oynar.[9] Uzun ve yapısal olarak düzensizliği olan CTD, transkripsiyon döngüsü sırasında fosforilasyona ve diğer post-translasyonel modifikasyonlara tabi olan çoklu heptapeptid sekansı YSPTSPS tekrarlarını içerir. Bu modifikasyonlar ve sebep oldukları düzenlemeleri, CTD'nin transkripsiyon başlangıcını, uzamasını ve sonlandırılmasını kontrol etmesi ve transkripsiyon ile RNA işlemesini kombinlemeyi sağlar
Başlama
Ökaryotlarda gen transkripsiyonunun başlatılması spesifik adımlarda gerçekleşir. İlk olarak, genel transkripsiyon faktörleriyle birlikte bir RNA polimeraz, ön başlama kompleksi olarak adlandırılan kapalı bir kompleks oluşturmak için genin promotör bölgesine bağlanır. Kompleksin daha sonra kapalı durumdan açık duruma geçişi, iki DNA şeridinin erimesine veya ayrılmasına ve kalıp ipliğin RNA polimerazının aktif bölgesine konumlandırılmasına neden olur. Bir primer gerekmeden, RNA polimeraz, ribonükleotit seçimini ve polimerizasyonu yönlendirmek için DNA sarmalını kullanarak yeni bir RNA zincirinin sentezini başlatabilir. Bununla birlikte, başlatılan sentezlerin çoğu, transkriptler önemli bir uzunluğa (-10 nükleotit) ulaşmadan önce durdurulur. Bu düşük döngüler sırasında, polimeraz, on nükleotidi aşan bir transkript üretene kadar kısa transkriptler üretmeye ve salmaya devam eder. Bu eşik elde edildiğinde, RNA polimeraz promotörü geçer ve transkripsiyon uzama fazına ilerler.

Ökaryotik promotörler ve genel transkripsiyon faktörleri
Pol II ile transkripsiyonu yapılmış genler, transkripsiyon başlangıç bölgesinin (TSS) hemen yakınında, başlangıç öncesi kompleksi bağlayan ve konumlandıran bir bölge içerir. Bu bölgeye, transkripsiyon başlangıcındaki önemli rolü nedeniyle çekirdek promotör adı verilir.[10][10] Promotörlerde farklı dizi elemanı sınıfları bulunur. Örneğin, TATA kutusu , birçok gende bağlanan transkripsiyon kompleksi montajını başlatan TATA kutusu bağlayıcı proteini olan TBP için oldukça korunmuş DNA tanıma dizisidir.
Ökaryotik genler ayrıca çekirdek promotörünün ötesinde düzenleyici sekanslar da içerir. Bu cis etkili kontrol elemanları , transkripsiyonu arttırmak veya azaltmak için çekirdek promotöründen transkripsiyonel aktivatörleri veya baskılayıcıları bağlar. İyi karakterize edilmiş düzenleyici öğeler arasında arttırıcılar , susturucular ve izolatörler bulunur . Bu düzenleyici diziler, çekirdek promotörlerden bazen yüzlerce kilobaz uzaktaki bir genomik mesafeye yayılabilir.
Genel transkripsiyon faktörleri, transkripsiyonun başlatılması ve düzenlenmesinde rol oynayan bir grup proteindir. Bu faktörler tipik olarak, çekirdek promotörün spesifik sekans elemanlarını bağlayan ve RNA polimerazının transkripsiyon başlangıç bölgesine çekilmesine yardımcı olan DNA-bağlama alanlarına sahiptir. RNA polimeraz II için genel transkripsiyon faktörleri TFIID , TFIIA , TFIIB , TTUF , TFnE ve TFIIH' dir.[10][10]
Ön hazırlık kompleksinin toplanması
Transkripsiyona hazırlamak için ~ 2 milyon dalton ön hazırlık kompleksi oluşturmak ve bunun için de çekirdek promotörde bir dizi genel transkripsiyon faktörü ve RNA polimerazının birleştirilmesi gerekir.[10] Örneğin, TSS'ye yakın bir TATA kutusu içeren promotörler için, TATA kutusunun TFIID'nin TBP alt birimi tarafından tanınması bir transkripsiyon kompleksinin montajını başlatır.Dahil olacak sonraki proteinler, DNA-TFIID kompleksini stabilize eden ve TFIIF ve ek transkripsiyon faktörleri ile birlikte Pol II'yi çağıran TFIIA ve TFIIB'dir. TFIIF, TATA'ya bağlı TBP ile polimeraz arasında köprü görevi görür. Ön hazırlık kompleksine dahil olacak son transkripsiyon faktörlerinden biri, promotörlerin erimesi ve kaçışında önemli bir rol oynayan TFIIH'dir.

Promotör erime ve açık kompleks oluşumu
Pol II ile transkribe edilen genler için ve bakteriyel RNA polimerazın aksine, promotör erimesi, ATP'nin hidrolizini gerektirir ve TFIIH'ye aracılık eder.[10] TFIIH, hem ATPaz hem de protein kinaz aktivitelerine sahipl olmakla beraber on alt birimli bir proteindir .[10] Yukarı akış promotörü DNA, TFIID tarafından sabit bir pozisyonda tutulurken, TFIIH aşağı akışlı çift sarmallı DNA'yı polimerazın yarığına çekerek DNA iplikçiklerinin ayrılmasını ve ön-hazırlık kompleksinin kapalı-açık durumları arasındaki geçişi sağlar. TFIIB, erimiş DNA'yı bağlayarak ve transkripsiyon baloncuğunu stabilize ederek açık kompleks oluşumuna yardımcı olur.
Abortif başlama
Başlatma kompleksi açıldığında, bir primer yokluğunda polimerizasyon reaksiyonunu başlatmak için birinci ribonükleotit aktif bölgeye getirilir. Bununla beraber, DNA ile bir hetero-dupleks oluşturan yeni oluşan bir RNA zinciri oluşturulmuş olunur. Uzama fazına girmeden önce, polimerazın işlevi erken sona erebilir ve kısa, kesik bir transkript salınabilir. Bu sürece abortif başlama denir. Transkript yeterli uzunlukta büyümeden önce promotörden polimeraz kaçışını teşvik etmek için birçok abortif başlama döngüsü meydana gelebilir. Abortif başlama döngüleri boyunca, RNA polimeraz promotere bağlı kalır ve aşağı akış DNA'sını bir çırpma-hareketi içinde katalitik yarığına çeker.[10]
Promotör kaçış
Bir transkript on nükleotitin eşik uzunluğuna ulaştığında, RNA çıkış kanalına girer. Polimeraz, promotör elementler ve başlangıç kompleksi ile bağlantılı herhangi bir düzenleyici protein ile etkileşimlerine artık ihtiyaç duymaz.[10] Ökaryotlarda promotör kaçış ATP hidrolizini ve CTD'nin Pol II-fosforilasyonunu gerektirir. Bu arada, transkripsiyon baloncuğu 12-14 nükleotite düşerek kaçış için gereken kinetik enerjiyi sağlar.
Uzama
Promotörden kaçtıktan ve başlatma için transkripsiyon faktörlerinin çoğunu attıktan sonra, polimeraz, transkripsiyonun bir sonraki aşaması olan uzama için yeni faktörler kazanır.[11][12] Transkripsiyon uzaması ilerleyici bir süreçtir. Enzimin önünden giren çift sarmallı DNA, RNA sentezi için kalıp sarmalın elde edilmesi için açılmıştır. İlerleyen polimeraz ile ayrılan her DNA baz çifti için, bir hibrit RNA: DNA baz çifti hemen oluşur. DNA zincirleri ve yeni RNA zinciri ayrı kanallardan çıkar; iki DNA zinciri, transkripsiyon kabarcığının arka ucunda bir araya gelirken, tek zincirli RNA tek başına ortaya çıkar.
Uzama faktörleri
Polimeraz için alınan proteinler arasında, uzatma faktörleri vardır, transkripsiyon uzamasını uyardıklarından bu ismi almıştır.[13] Farklı uzama faktörü sınıfları vardır. Bazı faktörler, genel olarak transkripsiyon oranını artırabilir, bazıları geçici duraklatma bölgelerinde polimeraza yardımcı olabilir ve bazıları polimerazın kromatinde transkripsiyonuna yardımcı olabilir.[14] Uzatma faktörlerinden biri olan P-TEFb, özellikle önemlidir.[15] P-TEFb, DNA'ya bağlı polimeraz II' nin CTD tekrarlarının (YSPTSPS) ikinci aminoasitini (Ser-2) fosforiller(fosforil ekler). P-TEFb aynı zamanda TAT-SF1 ve SPT5' i fosforiller ve aktive eder. SPT5, Ser-5'ten fosforile edilmiş CTD ile Pol II'ye 5' ucu kapaklama enziminin dahil edilmesine yardımcı olan evrensel bir transkripsiyon faktörüdür. TAF-SF1, RNA kesme sistemi bileşenlerini Ser-2 fosforile edilmiş CTD'ye dahil eder. P-TEFb ayrıca başlangıçtan hemen sonra belirli sekanslarla karşılaştığında geçici polimeraz duraklamasının bastırılmasına yardımcı olur.[15]
Transkripsiyon doğruluğu
Transkripsiyon doğruluğu çoklu mekanizmalarla elde edilir. RNA polimerazları, transkripsiyon hatalarını önlemek için doğru nükleosit trifosfat (NTP) substratını seçer. Yalnızca DNA'daki kodlama tabanıyla doğru şekilde eşleşen NTP, aktif merkeze kabul edilir.[16][17] RNA polimerazı yanlış belirlenmiş nükleotidleri tespit etmek ve kaldırmak için bilinen iki kontrol işlemi gerçekleştirir: pirofosforitik düzenleme ve hidrolitik düzenleme.[18] İlki, yanlış yerleştirilmiş ribonükleotidi, polimerizasyon reaksiyonunun basitçe tersine çevrilmesiyle ortadan kaldırırken, ikincisi, polimerazın geri çeker ve hata içeren RNA ürünü parçasının ayrılmasını içerir. Uzama faktörü TFIIS, polimerazda doğal olmayan bir ribonükleaz aktivitesini uyararak yanlış yerelleştirilmiş bazların sınırlı lokal RNA sindirimi yoluyla kurtulunmasına izin verir.[19] Tüm reaksiyonların (fosfodiester bağ sentezi, pirofosforroliz, fosfodiester bağ hidrolizi), tek bir aktif merkez kullanılarak RNA polimeraz ile yapılması dikkat çekicidir.[20]
Duraklatma, dengelenme ve geriye dönme
Transkripsiyon doğruluğu çoklu mekanizmalarla elde edilir. RNA polimerazları, transkripsiyon hatalarını önlemek için doğru nükleosit trifosfat (NTP) substratını seçer. Yalnızca DNA'daki kodlama tabanıyla doğru şekilde eşleşen NTP, aktif merkeze kabul edilNA polimerazı yanlış belirlenmiş nükleotidleri tespit etmek ve kaldırmak için bilinen iki kontrol işlemi gerçekleştirir: pirofosforitik düzenleme ve hidrolitik düzenleme.[21] İlki, yanlış yerleştirilmiş ribonükleotidi, polimerizasyon reaksiyonunun basitçe tersine çevrilmesiyle ortadan kaldırırken, ikincisi, polimerazın geri çeker ve hata içeren RNA ürünü parçasının ayrılmasını içerir. Uzama faktörü TFIIS, polimerazda doğal olmayan bir ribonükleaz aktivitesini uyararak yanlış yerelleştirilmiş bazların sınırlı lokal RNA sindirimi yoluyla kurtulunmasına izin verir.[19] Tüm reaksiyonların (fosfodiester bağ sentezi, pirofosforroliz, fosfodiester bağ hidrolizi), tek bir aktif merkez kullanılarak RNA polimeraz ile yapılması dikkat çekicidir.[20]
Duraklatma, dengelenme ve geriye dönme
Transkripsiyon uzaması, DNA dümdüz giden bir yol değildir. Düzeltme işlemi için polimeraz, yapılmış olan RNA'nın bazılarını silmek, yedeklemek ve başka bir transkripsiyona geçmek için yapılır.[22] Genel olarak, RNA polimeraz, bir gen boyunca sabit bir hızla transkripsiyon yapmaz. Aksine, belirli dizilerde, bazen de transkripsiyona devam etmeden önce uzun bir süre periyodik olarak duraklar.[23] Aşırı durumlarda, örneğin, polimeraz hasarlı bir nükleotitle karşılaştığında, tamamen durur. Daha sık olarak, uzayan bir polimeraz promotörün yakınında durur.[24] Uzamanın erken evreleri sırasında promoter-proksimal duraklatma, hızlı veya koordineli bir şekilde eksprese edilmek üzere dizaynı olan genleri düzenlemek için yaygın olarak kullanılan bir mekanizmadır. Duraklatma, DSIF (SPT4 / SPT5 içeren DRB-duyarlılık-indükleyici faktör/DRB-sensitivity-inducing factor) ile işbirliği içinde NELF (negatif uzama faktörü/negative elongation factor) olarak adlandırılan bir kompleksin aracılığı ile gerçekleşir.[25] Polimeraz, CTD kuyruğunun Ser-2'sinin P-TEFb tarafından fosforilasyonu gibi bir aktivasyon sinyali aldığında blokaj serbest bırakılır. ELL ve TFIIS gibi diğer uzama faktörleri, polimerazın duraklattığı süreyi sınırlayarak uzamayı tetikler.[26]
RNA işleme
Uzama sırasındaki haliyle polimeraz, çeşitli RNA işlem tipleri için gerekli olan bir dizi protein faktörü ile ilişkilidir. mRNA, polimerazın RNA çıkış kanalından çıktığı anda kapaklama işlemine maruz kalır. Kapaklamadan sonra, CTD tekrarları içindeki Ser-5'in fosforilasyonu, kapatma sistemlerinin ayrışmasından sorumlu olabilir. Ser-2'nin daha fazla fosforilasyonu, olgun mRNA üretmek için kodlamayan intronların çıkarılmasını katalize eden RNA kesme sistemlerinin işe alınmasına neden olur. Alternatif kesme, ökaryotlardaki protein komplemanlarını genişletir. Tıpkı 5’ ucun kapaklaması ve kesilmesinde olduğu gibi, CTD kuyruğu, transkripsiyonun sona ermesiyle meydana gelen son RNA işleme olayı olan 3' ucun poliadenilasyondan sorumlu olan enzimlerin toplanmasında rol oynar.[27]
Sonlanma
Transkripsiyonun son aşaması, sonlanmadır ve bu, tam transkriptin ayrılmasına ve RNA polimerazın şablon DNA'dan salınmasına yol açar. İşlem, üç RNA polimerazının her biri için farklıdır.[28] Sonlandırma mekanizması, üç transkripsiyon aşamasının en az anlaşılmış halidir.
Faktör bağımlı
Polimeraz Pol I ile pre-rRNA genlerinin transkripsiyonunun sonlandırılması, spesifik bir transkripsiyon sonlandırma faktörüne ihtiyaç duyan bir sistem tarafından gerçekleştirilir.[29] Kullanılan mekanizma prokaryotlarda rho bağımlı sonlandırmaya bazı benzerlikler taşımaktadır.[30] Ökaryotik hücreler, bazen çoklu kromozomlar üzerine dağıtılmış yüzlerce ribozomal DNA tekrarı içerir. Transkripsiyonun sonlandırılması, bir Pol I duraklatma bölgesinin yukarı akışında birkaç transkripsiyon sonlandırma bölgesi içeren ribozomal intergenik aralayıcı bölgede meydana gelir. Henüz bilinmeyen bir mekanizma sayesinde, transkriptin 3' ucu, olgun 18S, 5.8S ve 28S rRNA'larında ilerde işlenecek büyük bir primer rRNA molekülü oluşturarak ayrılır.
Pol II, bir genin sonuna ulaştığında, CTD, CPSF (bölünme ve poliadenilasyon spesifiklik faktörü/cleavage and polyadenylation specificity factor) ve CSTF (bölünme stimülasyon faktörü/cleavage stimulation factor) tarafından taşınan iki protein kompleksi, kopyalanan RNA'daki poli-A sinyalini tanır.[28] Poli-A bağlı CPSF ve CSTF, RNA' nın ayrılmasını sağlarve daha sonra poliadenilasyonu gerçekleştirmek için diğer proteinleri çağırır. Poli-A polimeraz RNA'nın ayrılmış 3' ucuna bir kalıp olmadan yaklaşık 200 adenin ekler.[28] Uzun poli-A kuyruğu Pol II tarafından yapılan transkriptlere özgüdür.
Pol I ve Pol II tarafından transkripsiyonun sonlandırılması sürecinde, uzama kompleksi, RNA ayrıldıktan hemen sonra çözülmez. Polimeraz, şablon boyunca ilerleyerek uzama kompleksi ile bağlantılı ikinci bir RNA molekülü oluşturur.[31] Sonlandırmanın nasıl başarıldığını açıklamak için iki model önerilmiştir.[28] Allosterik model, transkripsiyon sonlandırma sekansından geçtiğinde, uzama faktörlerinin sökülmesine ve / veya uzama kompleksinin konformasyonel değişikliklerine neden olan sonlandırma faktörlerinin bir araya getirilmesine neden olduğunu belirtir.[30][32] Torpido modeli, 5 'ila 3' eksonükleazının, uzama kompleksinden çıkan ikinci RNA'yı bozunduğunu göstermektedir. Polimeraz, son derece işlemsel eksonükleazın solladığı için salınır. Ortaya çıkan bir görüşün bu iki modelin birleşimini ifade edeceği ileri sürülmektedir.[32]
Faktör bağımsız
RNA polimeraz III, ek faktörlerin katılımı olmadan transkripsiyonu verimli bir şekilde sonlandırabilir. Pol III sonlandırma sinyali, olgun RNA'ların 3 'ucundan aşağı doğru 40bp içinde yer alan bir dizi timinlerden (kalıp olmayanzincirde bulunurlar) oluşur.[28] Poli-T sonlandırma sinyali Pol III'ü duraklatır ve en yakın RNA firketesine(saç tokası) geri dönmesine neden olur ve kompleks “kör uç” kompleks haline gelir.[33] Allosterik sonlandırma mekanizması ile tutarlı olarak ,[34] RNA firketesi Pol III'ü allosterik olarak açar ve uzama kompleksinin dağılmasına neden olur. Pol III-transkriptinde gömülü olan geniş yapı böylece Pol III'ün bir genin sonunda faktörden bağımsız olarak salınmasından sorumludur. RNA-dubleks bağımlı sonlandırma, son evrensel ortak ataya (last universal common ancestor/LUCA) dayanan eski bir mekanizmadır.
Ökaryotik transkripsiyonel kontrol
Ökaryotlarda gen ekspresyonunun düzenlenmesi, belirli bir hücresel ihtiyaca cevap olarak bireysel genleri açmak veya kapatmak için lokal olarak hareket eden ve genel olarak hücre kimliğini şekillendiren ve kromatin çapında gen ekspresyon modelini korumak için hareket eden birkaç kontrol seviyesinin etkileşimi yoluyla sağlanır.[35][36] Ökaryotik genom, nükleozomlar ve daha yüksek dereceli kromatin yapıları oluşturmak için histonların etrafına sarıldığından, transkripsiyonel sistemlerinin substratları genel olarak kısmen gizlenmiştir.[35] Düzenleyici proteinler olmadan, birçok gen düşük seviyede eksprese edilir veya hiç eksprese edilmez. Transkripsiyonel sistemlerinin DNA'ya erişmesini sağlamak için konumlandırılmış nükleozomların yer değiştirmesini gerektirir.[37]
Transkripsiyondaki tüm adımlar bir dereceye kadar düzenlemeye tabidir.[35] Özellikle transkripsiyon başlangıcı, gen ekspresyonunun düzenlendiği birincil seviyedir. Hız sınırlayıcı ilk adımı hedeflemek, hücre için enerji maliyetleri açısından en verimli olanıdır. Transkripsiyonun başlatılması, DNA'nın düzenleyici bölgelerinde bulunan cis etkili elemanlar (arttırıcılar, susturucular, izolatörler) ve aktivatörler veya baskılayıcılar olarak görev yapan sekansa spesifik transaktif faktörler tarafından düzenlenir.[35] Gen transkripsiyonu, uzamadaki polimerazın hareketini hedef alarak başlatma sonrası da düzenlenebilir.[38]
Global kontrol and epigenetik düzenleme
Ökaryotik genom, sadece DNA'ya düzenlenmiş erişime izin veren kompakt bir kromatin yapısı halinde yapılanmıştır. Kromatin yapısı genel olarak "açık" ve daha transkripsiyonel olarak izin verebilir veya genel olarak "yoğunlaştırılmış" ve transkripsiyonel olarak inaktif olabilir. İlki (ökromatin), aktif transkripsiyon altındaki genlerle doludur ve hafifçe paketlenmiştir. İkincisi (heterokromatin), telomerler ve sentromerler gibi genden fakir bölgeleri, fakat aynı zamanda normal gen yoğunluğuna sahip ancak transkripsiyonel olarak susturulmuş bölgeleri içerir. Transkripsiyon, histon modifikasyonu (deasetilleme ve metilasyon), RNA etkileşimi ve / veya DNA metilasyonu ile susturulabilir.[39]
Hücre kimliğini tanımlayan gen ekspresyon desenleri hücre bölünmesi yoluyla kalıtsaldır.[40] Bu işleme epigenetik düzenleme denir. DNA metilasyonu, replikasyon ile oluşturulan ortaya yeni çıkan DNA iplliğini modifiye eden bakım metilazlarının etkisiyle güvenilir bir şekilde kalıtsallaştırılır. Özelleşmiş proteinler işaretleyiciyi tanıyabilir ve susturmayı yeniden oluşturmak için histon deasetilazlar ve metilazları toplayabilir. Nükleozom histon modifikasyonları hücre bölünmesi sırasında da kalıtsal olabilir, ancak DNA metilasyonunun yönü olmadan bağımsız olarak çalışıp çalışamayacağı açık değildir.[40]
Gen spesifik aktivasyon
Transkripsiyonun başlatılmasının iki ana görevi, promotere erişimi olan RNA polimerazı sağlamak ve polimeraz içeren genel transkripsiyon faktörlerini bir transkripsiyon başlatma kompleksi içine monte etmektir. Gen promotöründe inhibe edici sinyalleri geçersiz kılarak transkripsiyonu başlatan farklı mekanizmalar tanımlanmıştır.[41] Ökaryotik genler, çok sayıda regülatör bağlama bölgesini kapsayan ve genel kilobazları (bazen yüzlerce kilobazı yayan) geniş düzenleyici sekanslar edinmiştir, promotörden - hem yukarı hem de aşağı yönde.[41] Regülatör bağlanma bölgeleri genellikle arttırıcılar denilen birimler halinde kümelenir. Arttırıcılar, bazı geliştiriciyi oluşturan birçok transkripsiyon faktörünün işbirliğine dayanan eylemini kolaylaştırabilir. Uzaktan geliştiriciler uzaktan transkripsiyon düzenlemesine izin verir. Güçlendiriciler ve destekleyiciler arasında yer alan izolatörler, bir geliştiricinin etkileyebileceği veya etkileyemediği genleri tanımlamaya yardımcı olur.
Ökaryotik transkripsiyonel aktivatörler, ayrı DNA bağlama ve aktivasyon fonksiyonlarına sahiptir.[42] Cis-elementine bağlandıktan sonra, bir aktivatör doğrudan polimeraz veya transkripsiyon sistemini tarafından ihtiyaç duyulan diğer faktörleri çağırabilir. Bir aktivatör ayrıca, promotörün yakınında kromatini değiştiren ve böylece başlatmaya yardımcı olan nükleozom değiştiricileri de işe alabilir. Çoklu aktivatörler, transkripsiyonel makinenin ortak veya iki karşılıklı bağımlı bileşenini işe alarak veya birbirlerine DNA sitelerine bağlanmalarına yardımcı olarak birlikte çağırabilirler.[43] Bu etkileşimler, hücresel ihtiyaçlara yönelik olarak çoklu sinyal girişlerini birleştirebilir ve karmaşık transkripsiyonel ürünler üretebilir.
Gen spesifik baskılama
Ökaryotik transkripsiyon baskılayıcıları prokaryotik meslektaşları tarafından kullanılan mekanizmaların bazılarını paylaşır. Örneğin, bir aktivatörün bağlanma bölgesi ile örtüşen DNA üzerindeki bir bölgeye bağlanarak bir baskılayıcı, aktivatörün bağlanmasını önleyebilir. Ancak, daha sık olarak, ökaryotik baskılayıcılar, aktifleştirici alanını maskeleyerek, nükleer lokalizasyonunu engelleyerek, bozulmasını teşvik ederek veya kimyasal modifikasyonlar yoluyla etkisiz hale getirerek bir aktivatörün işlevini engeller [44] Bir promotörün yukarısında bulunan bir bölge ile ve transkripsiyonel sistem ile etkileşime girerek bir baskılayıcı transkripsiyonun başlamasını direkt olarak engelleyebilir. Baskılayıcılar dolaylı olarak DNA'nın erişilebilirliğini etkileyen histon değiştiricileri (deasetilazlar ve metilazlar) veya nükleozom yeniden modelleme enzimlerini çağırarak transkripsiyonu bastırabilirler.[45] DNA ve histon değişikliklerini baskılamak kromatin boyunca yayılacak ve çoklu genleri kapatacak transkripsiyonel susturmanın temelidir.[46]
Uzama ve sonlanmanın kontrolü
Uzama fazı, uzatma kompleksinin montajı tamamlandıktan sonra başlar ve bir sonlandırma sekansı ile karşılaşılana kadar ilerler.[47] RNA polimerazının başlatılmasından sonraki hareketi, önemli düzenleyici mekanizmaların başka bir sınıfının hedefidir. Örneğin, transkripsiyonel aktivatör Tat, HIV transkripsiyonunun düzenlenmesi sırasında başlamayı değil, uzamayı etkiler.[48] Aslında, birçok ökaryotik gen, promotör-proksimal duraklatma olarak adlandırılan transkripsiyon uzamasına bir blok bırakılarak düzenlenir.[49] Duraklatma, gen aktivitesini kolaylaştırmak ve hücreler bir aktivasyon sinyaline maruz kaldığında hızlı veya senkronize transkripsiyonel tepkilere yol açmak için promotörlerde kromatin yapısını etkileyebilir.[50] Duraklatma, iki negatif uzatma faktörünün (DSIF (SPT4 / SPT5) ve NELF), uzama kompleksine bağlanması ile ilişkilidir. Diğer faktörler ayrıca duraklatılmış polimerazın stabilitesini ve süresini de etkileyebilir.[51] Duraklama serbest bırakması, P-TEFb kinazının alımıyla tetiklenir.[52]
Transkripsiyon sonlandırma ayrıca, transkripsiyonel regülasyonun önemli bir alanı olarak ortaya çıkmıştır. Sonlandırma polimerazın verimli geri dönüşümü ile birleştirilir.[53] Transkripsiyon sonlandırma ile ilişkili faktörler ayrıca gen döngüsüne aracılık eder ve böylece yeniden başlatmanın etkinliğini belirler.
Transkripsiyon-eşlik eden DNA onarımı
Transkripsiyon, bir genin transkripsiyonunda bulunan bir lezyonun varlığı ile tutuklu kaldığında, DNA onarım proteinleri, transkripsiyona eşlik eden onarım olarak adlandırılan bir işlemi başlatmak için duraklatılmış RNA polimerazına alınır.[54] Bu sürecin merkezinde, ATPase aktivitesine sahip olan genel transkripsiyon faktörü TFIIH vadır. TFIIH, DNA tamir enzimlerinin lezyona erişebilmesi için içeride kalmış olan transkripsiyon balonunu açığa çıkarmak amacıyla polimerazda bir konformasyonel değişikliğe neden olur.[55] Bu nedenle, RNA polimeraz, tamir enzimlerini aktif olarak kopyalanan genlere hedeflemek için hücrede hasar algılayıcı protein görevi görür.
Prokaryotik ve ökaryotik transkripsiyon arasındaki karşılaştırmalar
Ökaryotik transkripsiyon prokaryotik transkripsiyondan daha karmaşıktır. Örneğin, ökaryotlarda genetik materyal (DNA) ve dolayısıyla transkripsiyon, esas olarak, nükleer membran tarafından sitoplazmadan (translasyonun meydana geldiği) ayrıldığı çekirdeğe yerleşiktir. Bu, gen ekspresyonunun çekirdekteki RNA'nın hapsi yoluyla geçici olarak düzenlenmesini sağlar ve olgun RNA'ların sitoplazmaya seçici olarak taşınmasını sağlar. Bakteriler, DNA'yı ribozomdan ayıran ayrı bir çekirdeğe sahip değildir ve mRNA, kopyalandığı anda proteine çevrilir. İki işlem arasındaki eşleşme, prokaryotik gen regülasyonu için önemli bir mekanizma sağlar.[56]
Başlama adımında, prokaryotlardaki RNA polimeraz (özellikle bakteriler) promotör bölgeye kuvvetli bir şekilde bağlanır ve yüksek bir bazal transkripsiyon oranı başlatır. Kapalıdan açığa geçiş için ATP hidrolizine gerek yoktur, promotör eritmesi erimiş konformasyonu destekleyen bağlanma reaksiyonları ile tahrik edilir. Kromatin ökaryotlarda transkripsiyonu büyük ölçüde engeller. Promotere özgü başlatma için büyük çoklu protein ön hazırlama kompleksinin birleştirilmesi gerekir. Ökaryotlarda eriyen promotör, ATP'nin hidrolizini gerektirir. Sonuç olarak, ökaryotik RNA polimerazları düşük bir bazal transkripsiyon başlatma hızı sergilemektedir.[57]
Kanserde transkripsiyonun düzenlenmesi
Omurgalılarda, gen promotörlerinin çoğu, çok sayıda CpG bölgesine sahip bir CpG adası içerir.[58] Bir genin promotör CpG bölgelerinin çoğu metillendiğinde gen susturulur.[59] Kolorektal kanserler tipik olarak 3 ila 6 sürücü mutasyonuna ve 33 ila 66 otostopçu veya yolcu mutasyonuna sahiptir.[60] Bununla birlikte, transkripsiyonel susturma, kansere doğru ilerlemede mutasyondan daha önemli olabilir. Örneğin, kolorektal kanserlerde, yaklaşık 600 ila 800 gen, CpG adası metilasyonu ile transkripsiyonel olarak susturulur (bakınız kanserde transkripsiyonun düzenlenmesi). Kanserde transkripsiyonel baskılama, mikroRNA' ların değişmiş ekspresyonu gibi diğer epigenetik mekanizmalar tarafından da oluşabilir.[61] Meme kanserinde, aşırı eksprese edilen mikroRNA-182 aracılığı ile BRCA1'in transkripsiyonel baskılanması, BRCA1 promoterinin hipermetilasyonuyla basklamadan daha sık meydana gelebilir (bakınız meme ve yumurtalık kanserlerinde BRCA1'in düşük ekspresyonu).
Ayrıca bakınız
- Bakteriyel transkripsiyon
- Gen ekspresyonunun düzenlenmesi
- RNA polimeraz
- Transkripsiyonel düzenleme
- Transkripsiyon faktörü
Kaynakça
- Watson, James; Tania A. Baker; Stephen P. Bell; Alexander Gann; Michael Levine; Richard Losik; Stephen C. Harrison. Molecular Biology of the Gene (7th bas.). Benjamin-Cummings Publishing Company. ISBN 978-0-321-76243-6.
- Mattick, J. S. (1 Kasım 2001). "Non-coding RNAs: the architects of eukaryotic complexity". EMBO Reports. 2 (11). ss. 986-991. doi:10.1093/embo-reports/kve230. PMC 1084129 $2. PMID 11713189.
- Sirri, Valentina; Silvio Urcuqui-Inchima; Pascal Roussel; Danièle Hernandez-Verdun (2008). "Nucleolus: the fascinating nuclear body". Histochem. Cell Biol. 129 (1). ss. 13-31. doi:10.1007/s00418-007-0359-6. PMC 2137947 $2. PMID 18046571.
- Fromont-Racine, Micheline; Senger, Bruno; Saveanu, Cosmin; Fasiolo, Franco (Ağustos 2003). "Ribosome assembly in eukaryotes". Gene. Cilt 313. ss. 17-42. doi:10.1016/S0378-1119(03)00629-2.
- Dieci, Giorgio; Fiorino, Gloria; Castelnuovo, Manuele; Teichmann, Martin; Pagano, Aldo (Aralık 2007). "The expanding RNA polymerase III transcriptome". Trends in Genetics. 23 (12). ss. 614-622. doi:10.1016/j.tig.2007.09.001. PMID 17977614.
- Carter, R.; Drouin, G. (21 Aralık 2009). "The Increase in the Number of Subunits in Eukaryotic RNA Polymerase III Relative to RNA Polymerase II Is due to the Permanent Recruitment of General Transcription Factors". Molecular Biology and Evolution. 27 (5). ss. 1035-1043. doi:10.1093/molbev/msp316. PMID 20026480.
- Fernández-Tornero, Carlos; Moreno-Morcillo, María; Rashid, Umar J.; Taylor, Nicholas M. I.; Ruiz, Federico M.; Gruene, Tim; Legrand, Pierre; Steuerwald, Ulrich; Müller, Christoph W. (23 Ekim 2013). "Crystal structure of the 14-subunit RNA polymerase I". Nature. 502 (7473). ss. 644-649. doi:10.1038/nature12636.
- Cramer, P. (19 Nisan 2001). "Structural Basis of Transcription: RNA Polymerase II at 2.8 Angstrom Resolution". Science. 292 (5523). ss. 1863-1876. doi:10.1126/science.1059493. PMID 11313498.
- Corden, Jeffry L. (13 Kasım 2013). "RNA Polymerase II C-Terminal Domain: Tethering Transcription to Transcript and Template". Chemical Reviews. 113 (11). ss. 8423-8455. doi:10.1021/cr400158h. PMC 3988834 $2. PMID 24040939.
- Boş kaynak (yardım)
- Pokholok, Dmitry K; Hannett, Nancy M; Young, Richard A (Nisan 2002). "Exchange of RNA Polymerase II Initiation and Elongation Factors during Gene Expression In Vivo". Molecular Cell. 9 (4). ss. 799-809. doi:10.1016/S1097-2765(02)00502-6. PMID 11983171.
- Wade, Joseph T; Struhl, Kevin (Nisan 2008). "The transition from transcriptional initiation to elongation". Current Opinion in Genetics & Development. 18 (2). ss. 130-136. doi:10.1016/j.gde.2007.12.008. PMC 2563432 $2. PMID 18282700.
- Saunders, Abbie; Core, Leighton J.; Lis, John T. (Ağustos 2006). "Breaking barriers to transcription elongation". Nature Reviews Molecular Cell Biology. 7 (8). ss. 557-567. doi:10.1038/nrm1981. PMID 16936696.
- Arndt, Karen; Caroline Kane (1 Ekim 2003). "Running with RNA polymerase: eukaryotic transcript elongation". Trends in Genetics. 19 (10). ss. 543-550. doi:10.1016/j.tig.2003.08.008. PMID 14550628.
- Brès, Vanessa; Yoh, Sunnie M; Jones, Katherine A (Haziran 2008). "The multi-tasking P-TEFb complex". Current Opinion in Cell Biology. 20 (3). ss. 334-340. doi:10.1016/j.ceb.2008.04.008. PMC 2628440 $2. PMID 18513937.
- Westover, Kenneth D.; Bushnell, David A.; Kornberg, Roger D. (Kasım 2004). "Structural Basis of Transcription". Cell. 119 (4). ss. 481-489. doi:10.1016/j.cell.2004.10.016. PMID 15537538.
- Wang, Dong; Bushnell, David A.; Westover, Kenneth D.; Kaplan, Craig D.; Kornberg, Roger D. (Aralık 2006). "Structural Basis of Transcription: Role of the Trigger Loop in Substrate Specificity and Catalysis". Cell. 127 (5). ss. 941-954. doi:10.1016/j.cell.2006.11.023. PMC 1876690 $2. PMID 17129781.
- Wang, D.; Bushnell, D. A.; Huang, X.; Westover, K. D.; Levitt, M.; Kornberg, R. D. (28 Mayıs 2009). "Structural Basis of Transcription: Backtracked RNA Polymerase II at 3.4 Angstrom Resolution". Science. 324 (5931). ss. 1203-1206. doi:10.1126/science.1168729. PMC 2718261 $2. PMID 19478184.
- Sosunov, Vasily; Ekaterina Sosunova; Arkady Mustaev; Irina Bass; Vadim Nikiforov; Alex Goldfarb (2003). "Unified two-metal mechanism of RNA synthesis and degradation by RNA polymerase". The EMBO Journal. 22 (9). ss. 2234-2244. doi:10.1093/emboj/cdg193. PMC 156065 $2. PMID 12727889.
- Adelman, Karen; Lis, John T. (18 Eylül 2012). "Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans". Nature Reviews Genetics. 13 (10). ss. 720-731. doi:10.1038/nrg3293. PMC 3552498 $2. PMID 22986266.
- Missra, A.; Gilmour, D. S. (4 Haziran 2010). "Interactions between DSIF (DRB sensitivity inducing factor), NELF (negative elongation factor), and the Drosophila RNA polymerase II transcription elongation complex". Proceedings of the National Academy of Sciences. 107 (25). ss. 11301-11306. doi:10.1073/pnas.1000681107. PMC 2895096 $2. PMID 20534440.
- Richard, P.; Manley, J. L. (1 Haziran 2009). "Transcription termination by nuclear RNA polymerases". Genes & Development. 23 (11). ss. 1247-1269. doi:10.1101/gad.1792809. PMC 2763537 $2. PMID 19487567.
- Clancey, Suzanne (2008). "DNA transcription". Nature Education. 1 (41). 3 Aralık 2013 tarihinde kaynağından arşivlendi. Erişim tarihi: 27 Kasım 2013.
- Rosonina, E. (1 Mayıs 2006). "Terminating the transcript: breaking up is hard to do". Genes & Development. 20 (9). ss. 1050-1056. doi:10.1101/gad.1431606. PMID 16651651.
- Nielsen, S.; Yuzenkova, Y.; Zenkin, N. (27 Haziran 2013). "Mechanism of Eukaryotic RNA Polymerase III Transcription Termination". Science. 340 (6140). ss. 1577-1580. doi:10.1126/science.1237934. PMC 3760304 $2. PMID 23812715.
- Epshtein, Vitaly; Cardinale, Christopher J.; Ruckenstein, Andrei E.; Borukhov, Sergei; Nudler, Evgeny (Aralık 2007). "An Allosteric Path to Transcription Termination". Molecular Cell. 28 (6). ss. 991-1001. doi:10.1016/j.molcel.2007.10.011. PMID 18158897.
- Shandilya, J; Robert SG (2012). "The transcription cycle in eukaryotes: from productive initiation to RNA polymerase II recycling". Biochim Biophys Acta. 1819 (5). ss. 391-400. doi:10.1016/j.bbagrm.2012.01.010. PMID 22306664.
- Kulaeva, Olga; Daria Gaykalova; Vasily M. Studitsky (2007). "Transcription Through Chromatin by RNA polymerase II: Histone Displacement and Exchange". Mutat. Res. 618 (1–2). ss. 116-129. doi:10.1016/j.mrfmmm.2006.05.040. PMC 1924643 $2. PMID 17313961.
- Peterlin, BM; DH Price (2006). "Controlling the elongation phase of transcription with P-TEFb". Molecular Cell. 23 (3). ss. 297-305. doi:10.1016/j.molcel.2006.06.014. PMID 16885020.
- Bannister, Andrew J.; Kouzarides, Tony (15 Şubat 2011). "Regulation of chromatin by histone modifications". Cell Research. 21 (3). ss. 381-395. doi:10.1038/cr.2011.22. PMC 3193420 $2. PMID 21321607.
- Brown, Terrance A. (2002). Genomes. New York: Wiley-Liss. ISBN 978-0-471-25046-3.
- Kao, Shaw-Yi; Andrew Calman; Paul Luciw; Matija Peterlin (3 Aralık 1987). "Anti-termination of transcription within the long terminal repeat of HIV-1 by tat gene product". Nature. 330 (6147). ss. 489-493. doi:10.1038/330489a0. PMID 2825027.
- Lis, J (1998). "Promoter-associated Pausing in Promoter Architecture and Postinitiation Transcriptional Regulation". Cold Spring Harb Symp Quant Biol. Cilt 63. ss. 347-56. doi:10.1101/sqb.1998.63.347. PMID 10384299.
- Cheng B, Li T, Rahl PB, Adamson TE, Loudas NB, Guo J, Varzavand K, Cooper JJ, Hu X, Gnatt A, Young RA, Price DH (Ocak 2012). "Functional Association of Gdown1 with RNA Polymerase II Poised on Human Genes". Molecular Cell. 45 (1). ss. 38-50. doi:10.1016/j.molcel.2011.10.022. PMC 3259526 $2. PMID 22244331.
- Feige, MJ; LM Hendershot (Nisan 2011). "Disulfide bonds in ER protein folding and homeostasis". Curr Opin Cell Biol. 23 (2). ss. 167-75. doi:10.1016/j.ceb.2010.10.012. PMC 3078216 $2. PMID 21144725.
- MELLON, I (Ekim 1987). "Selective removal of transcription-blocking DNA damage from the transcribed strand of the mammalian DHFR gene". Cell. 51 (2). ss. 241-249. doi:10.1016/0092-8674(87)90151-6.
- Sarker, Altaf H.; Tsutakawa, Susan E.; Kostek, Seth; Ng, Cliff; Shin, David S.; Peris, Marian; Campeau, Eric; Tainer, John A.; Nogales, Eva; Cooper, Priscilla K. (Ekim 2005). "Recognition of RNA Polymerase II and Transcription Bubbles by XPG, CSB, and TFIIH: Insights for Transcription-Coupled Repair and Cockayne Syndrome". Molecular Cell. 20 (2). ss. 187-198. doi:10.1016/j.molcel.2005.09.022. PMID 16246722.
- Saxonov S, Berg P, Brutlag DL (2006). "A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters". Proc. Natl. Acad. Sci. U.S.A. 103 (5). ss. 1412-7. doi:10.1073/pnas.0510310103. PMC 1345710 $2. PMID 16432200.
- Bird A (2002). "DNA methylation patterns and epigenetic memory". Genes Dev. 16 (1). ss. 6-21. doi:10.1101/gad.947102. PMID 11782440.
- Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (2013). "Cancer genome landscapes". Science. 339 (6127). ss. 1546-58. doi:10.1126/science.1235122. PMC 3749880 $2. PMID 23539594.
- Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, Vecchiotti D, Capece D, Zazzeroni F, Alesse E (2014). "MicroRNAs in the DNA Damage/Repair Network and Cancer". Int J Genom. Cilt 2014. ss. 1-10. doi:10.1155/2014/820248. PMC 3926391 $2. PMID 24616890.